El descubrimiento de la estructura del ácido desoxirribonucleico (ADN) en 1953 por los científicos Francis Crick, de Gran Bretaña y James Watson, de Estados Unidos fue considerado un avance histórico para la ciencia, debido a que este hallazgo sentó las bases para la creación de nuevas disciplinas científicas como la biología molecular, la proteómica, metabolómica, farmacogenómica, entre muchas otras.
Gracias a este descubrimiento, hoy en día sabemos que aquella molécula químicamente sencilla, de doble hélice (parecida a una escalera de caracol) compuesta de cuatro bases (Adenina, Guanina, Citosina y Timina) que se combinan entre sí y repiten millones de veces guardan la información genética necesaria para el desarrollo y funcionamiento de los seres vivos.
Lograr descifrar hace 50 años cómo se acomoda esta molécula espacialmente no fue una tarea fácil de realizar, sin embargo, después de las aportaciones de diversos científicos de esa época, se llegó a la construcción física del modelo tridimensional que en aquel tiempo fue ensamblado pieza por pieza como si se tratase de un rompecabezas. En la actualidad, repetir esa hazaña en cada uno de los experimentos representaría una gran inversión de tiempo y recursos, es ahí donde toma su papel el uso de la bioinformática.
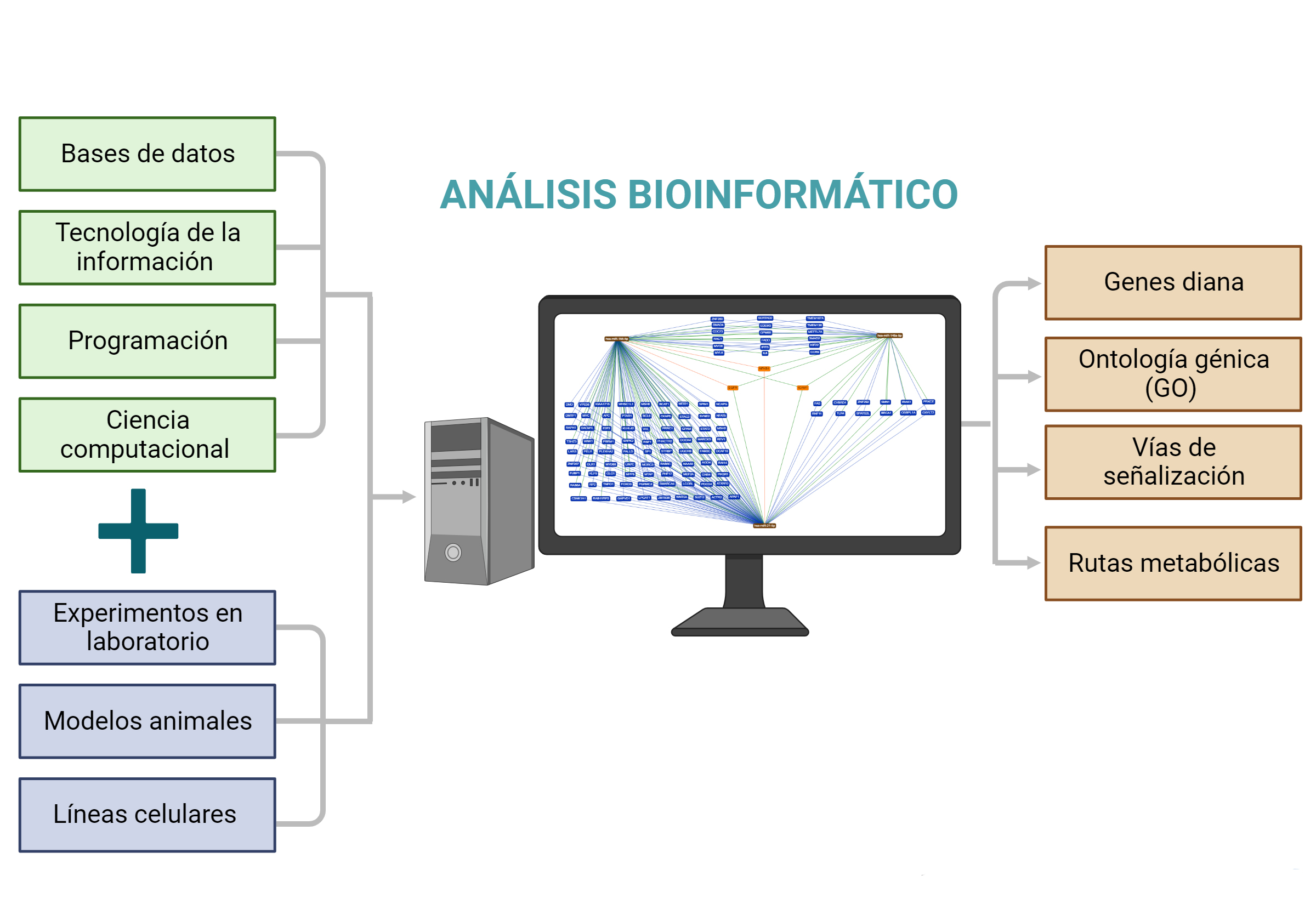
La bioinformática en palabras sencillas es el estudio de la información biológica en la que se combinan varias disciplinas como lo es la biología, la computación y las matemáticas. Esta disciplina tiene por objetivo adquirir, almacenar, analizar e interpretar la enorme cantidad de información y de datos provenientes del proyecto del genoma humano y que, a su vez, se ha extendido a otros genomas de organismos como animales o plantas (Gauthier, Vincent et al. 2019). Algunas de las metas que podemos destacar de esta disciplina son:
• Almacenar la información obtenida en una base de datos para que posteriormente sea organizada y analizada de manera más sencilla por diferentes usuarios.
• Generar modelos gráficos o numéricos que permitan predecir cambios que podrían llegar a sufrir ciertos datos en el futuro (esto se asemeja a una bola de cristal).
• Procesar la información contenida en los datos mediante el empleo de software de tal forma que sea más sencilla de analizar e interpretar (especialmente útil para aquellos que no son especialistas en el área).
Son diversas las aplicaciones de la bioinformática, por ejemplo, en medicina; ha permitido la medicina personalizada, es decir, la adaptación de tratamientos médicos específicos en cada persona, tomando en cuenta su genética; el uso de esta herramienta permitió el desarrollo en tiempo récord de la vacuna contra el virus SARS-CoV-2 que dejó millones de muertos a consecuencia de la enfermedad por COVID-19 (Ma, Li et al. 2021), en la agricultura; permite el desarrollo de cultivos que resistan a plagas y sequías prolongadas, y ambientalmente; permite la identificación de microorganismos con ciertas características que ayudan a la limpieza de aguas residuales o en su defecto, la eliminación de sustancias tóxicas del ambiente. En este sentido, también ha impactado en áreas que últimamente han tomado importante relevancia como lo es la toxicología.
Es así como en el Laboratorio de Toxicología Molecular del Centro de Investigación Aplicada en Ambiente y Salud (CIAAS) perteneciente a la Coordinación para la Innovación y Aplicación de la Ciencia y Tecnología (CIACYT) ha comenzado a aplicar esta innovadora herramienta. Nuestro grupo de investigación realizó un estudio (Fernández-Macias et al. 2023) en donde células mononucleares de sangre periférica (PBMC) fueron expuestas a diclorodifeniltricloroetano (DDT), un pesticida que se usó ampliamente para el control de plagas en la agricultura y/o que portaban enfermedades como el paludismo (malaria), pero que tiempo después fue prohibido porque se descubrió que podía causar efectos nocivos para la salud. El objetivo del estudio realizado por el grupo de Toxicología Molecular fue identificar mediante análisis bioinformático la red de genes y vías de señalización reguladas por el insecticida en células sanguíneas humanas expuestas al DDT en una investigación “in vitro”.
Entre los resultados más destacados en este estudio fueron los siguientes: el software bioinformático mostró que en conjunto se modularon 114 genes. Adicionalmente, el análisis también arrojó información acerca de las vías de señalización relacionadas con los genes modulados, entre las que se destacan vías de señalización asociadas a cáncer, muerte celular, ciclo celular, metabolismo, inflamación, entre otros.
El empleo de esta herramienta en los laboratorios de investigación básica abre nuevas puertas hacia la información genómica y permite que los investigadores incluso con poco adiestramiento informático utilicen toda la información disponible para complementar sus trabajos y así encontrar respuestas a las nuevas incógnitas que se generan día a día.
Referencias:
Carlos, F. J., T. C. Gabriel, P. A. Genoveva, V. J. Antonio and P. I. Nelinho (2023). "Expression levels and network analysis of inflammamiRs in peripheral blood mononuclear cells exposed to DDE "in vitro"." Environ Toxicol Pharmacol 97: 104032.
Gauthier, J., A. T. Vincent, S. J. Charette and N. Derome (2019). "A brief history of bioinformatics." Brief Bioinform 20(6): 1981-1996.
Ma, L., H. Li, J. Lan, X. Hao, H. Liu, X. Wang and Y. Huang (2021). "Comprehensive analyses of bioinformatics applications in the fight against COVID-19 pandemic." Comput Biol Chem 95: 107599.
